2023年4月12日,中国科学院北京生命科学研究院赵方庆团队在Nature Protocols杂志上发表了题为“Full-length circular RNA profiling by nanopore sequencing with CIRI-long”的文章,建立了环形RNA全长转录本解析和高效挖掘的技术体系。
近年研究表明,环形RNA在真核生物体内具有广泛的生物学功能。由于环形RNA序列与线性RNA相似度极高,因此如何高效鉴定环形RNA特有的外显子结构,一直是环形RNA研究中的重要问题。针对该问题,赵方庆团队前期提出了CIRI-AS算法,基于BSJ读段对比结果对环形RNA内部可变剪接结构进行识别(Genome Biology, 2015;Nature Communications, 2016)。后续研究中,研究组开发了CIRI-full算法,通过识别双端250bp测序数据中反向重叠区特征,对500bp以内的环形RNA进行了全长重构(Genome Medicine, 2019;Cell Reports, 2019;Nature Communications, 2020)。由于上述方法主要基于短读长测序技术,难以对长度500bp以上的环形RNA的全长序列进行有效识别。在此基础上,研究组开发了基于纳米孔测序的环形RNA全长测定方法CIRI-long,实现了对不同长度环形RNA全长结构的高效鉴定(Nature Biotechnology, 2021)。
在该研究中,赵方庆团队利用小鼠脑组织及人HeLa细胞系样本作为示例,详细描述了环形RNA高效富集与全长测定方法:首先,利用核糖体RNA去除试剂盒与改进的RNase R处理流程,消化RNA样本中的核糖体RNA与其余线性RNA分子,对环形RNA进行初步富集;随后,利用随机引物逆转录与PCR扩增,构建cDNA文库。该过程中,环形RNA模板可以发生滚环逆转录,产生含有多个环形RNA全长序列的嵌合体cDNA,而来自线性RNA的cDNA产物长度较短。因此,后续流程中使用了片段筛选策略,对环形RNA来源的cDNA片段进行了进一步富集,从而有效提升了文库中环形RNA来源片段所占比例。最后,利用长读长纳米孔测序技术进行cDNA分子的全长测定,并使用该团队开发的CIRI-long算法,对测序数据中的环形RNA序列进行识别与错误校正,从而获得高置信度的环形RNA全长结构。
在研究团队前期工作中,观察到使用具有模板切换活性的逆转录酶,可以在滚环逆转录产生的cDNA末端直接引入PCR引物序列,从而实现对低起始量环形RNA的高效捕获(图1)。然而由于该活性依赖5’ RNA线性末端参与,因此不含5’与3’线性末端的环形RNA如何发生模板切换的原理仍尚未明确。该论文中,作者进一步利用体外转录构建的模式环形RNA对滚环逆转录过程进行了进一步探究,发现经过逆转录后,大部分环形RNA模板发生了水解,从而提供了模板切换所需的线性5’末端。同时,在不加入逆转录酶的对照组中,环形RNA均保持了完整的环状结构。该结果提示环形RNA滚环反转录中的模板切换主要依赖RNA模板的线性化水解,为CIRI-long流程提供了重要的理论支持。
同时,该研究对CIRI-long方法的应用进行了总结,指出CIRI-long方法可以为新型环形RNA,如线粒体来源环形RNA或内含子自连型环形RNA的存在提供有力的支持证据。同时,重构获得的环形RNA全长序列可以用于后续环形RNA的功能预测(如miRNA或RBP结合位点分析)。另外,利用长读长测序方法构建出的环形RNA序列,可作为评估基于传统二代测序技术的环形RNA拼接算法的标准数据集,为现有算法的改进提供指导方向。与近年开发的多个长读长环形RNA测序技术相比,CIRI-long方法实验步骤更为便捷,同时可以实现对不同长度环形RNA全长结构的直接测定,可以应用于多种组织与细胞样本,具有广泛的应用前景。
该研究由中科院北京生命科学研究院赵方庆研究员团队完成,并获得了国家自然科学基金委杰青项目和重点项目的资助。
原文链接:https://www.nature.com/articles/s41596-023-00815-w
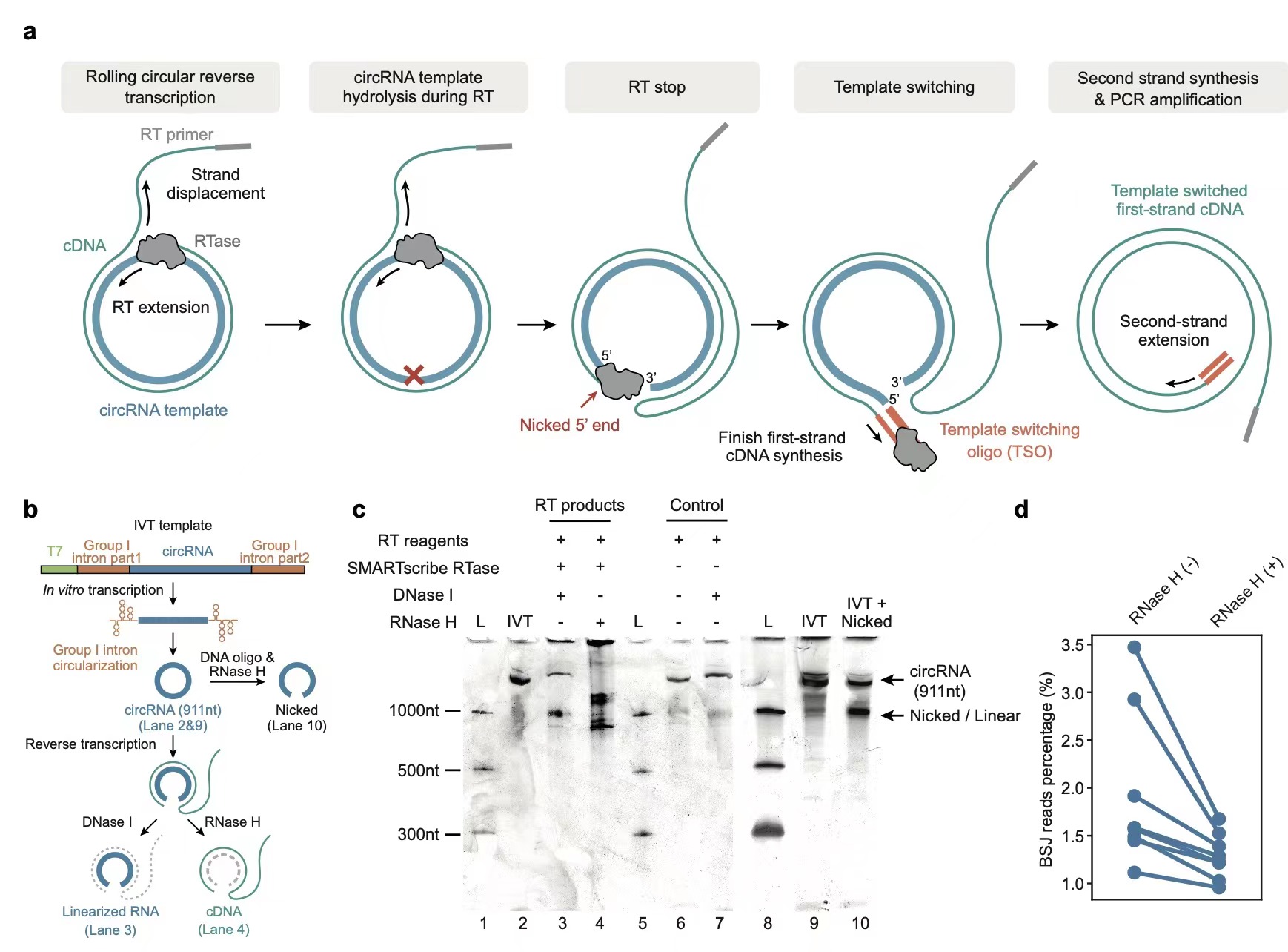
图1. 环形RNA滚环扩增及验证体系

