绝大部分遗传变异(尤其是同义突变)或RNA编辑并不会引起蛋白结构的改变,因此,这些遗传变异或RNA编辑是如何影响生物表型的尚不清楚。RiboSNitches是可以改变RNA结构的单核苷酸变异体(SNVs),它的丢失可能会导致特定RNA结构的改变,这种现象发生在成千上万的区域位点,其中包括3'-UTRs、microRNA的结合位点、RNA结合蛋白(RBPs)的结合位点。因此评估SNVs对转录后调控网络的作用将有助于理解不引起蛋白结构改变的遗传变异或RNA编辑影响生物表型的机制。
近日,北京生科院孙中生课题组开发了一个研究转录后因子互作和调控功能变异的数据库——RBP-Var(http://www.rbp-var.biols.ac.cn/)。RBP-Var提供了一个易于使用的Web界面,它能帮助用户快速判断自己感兴趣的SNVs是否可以改变RNA二级结构,并判断RBPs结合是否随后被破坏。RBP-Var可以评估每个SNV对miRNA-RNA相互作用的影响,因为SNVs可能会破坏或创建miRNA结合位点,最终导致丧失功能和/或获得性功能的miRNA -RNA相互作用。此外,通过建立启发式分类算法,RBP-Var可以评估遗传突变或RNA编辑对转录后调控网络的全局影响及其权重。总之,RBP-Var通过整合DNA和RNA生物学来解释遗传变异和转录后调控机制是如何协作并调控基因表达的,为筛选候选SNVs开展进一步的功能研究和探索人类疾病的潜在致病SNVs提供了一个行之有效的方法。
以上研究结果于2015年12月在线发表于Nucleic Acids Research期刊上(http://nar.oxfordjournals.org/content/early/2015/12/03/nar.gkv1308.long),此工作是博士生毛凤彪在孙中生研究员的指导下完成的。此项工作得到了国家高技术研究发展计划(863计划)、国家自然科学基金委和阿斯利康中国创新研究中心等项目的资助。
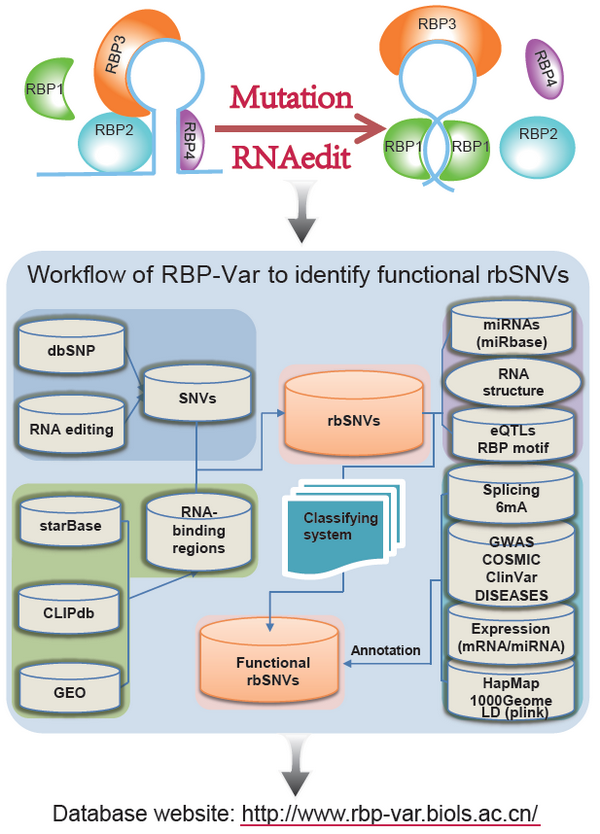
RBP-Var数据分析流程图

