近日,国际学术期刊Trends in Genetics在线发表了中国科学院北京生命科学研究院计算基因组学实验室赵方庆研究员的“Computational strategies for exploring circular RNAs”的综述文章。该论文全面阐述了环形RNA研究和数据挖掘中诸多方法,探讨了相关方法在非编码RNA数据挖掘中的适用条件与优劣评估,并指出未来环形RNA数据挖掘的发展趋势与挑战。
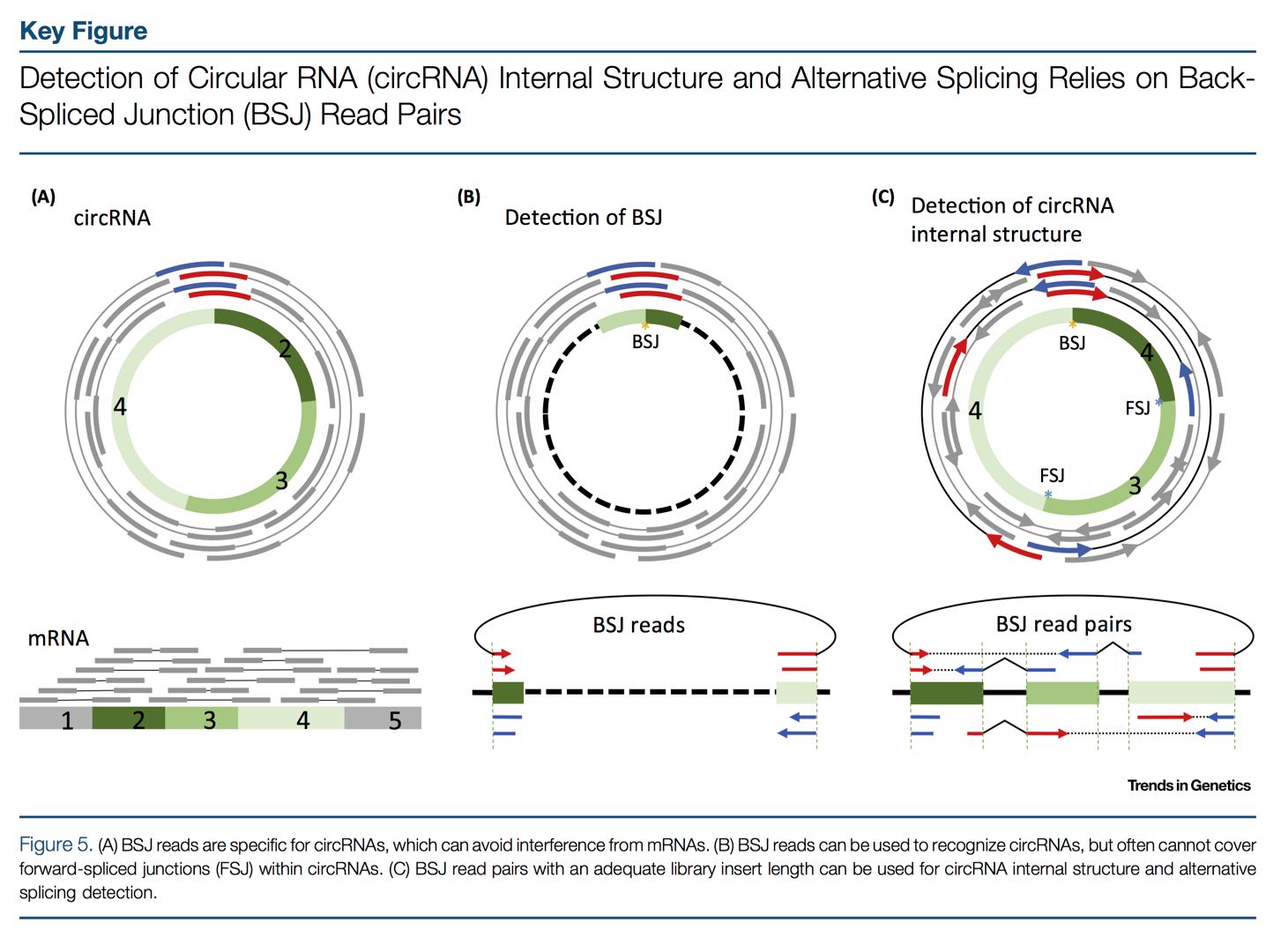
环形RNA是近年来获得广泛关注的一类结构呈闭合环形的RNA分子,并入选Clarivate Analytics 2017年度热点前沿领域。环形RNA的基因来源、内部组成、细胞定位、生成机制与生物功能均较为多样,通过高通量测序数据的挖掘对其深入研究成为该领域的必经途径。依据参考基因组的使用策略,现有的识别算法可划分为基于分段比对(split-alignment based)和基于伪参考序列构建(pseudo-reference based)两大类。由于所借助比对算法类型的不同,各识别算法又分别针对剪切型(splice-aware)和全能型(versatile)比对算法进行优化。此外,在向后剪接读段(back-spliced junction read)的检测和配对末端比对信息的筛选上,这些识别算法采用的策略也不尽相同。以上关键步骤极大影响了识别算法在不同转录组测序数据上的表现,目前现有的十余种环形RNA识别算法在敏感度、可靠性和适用范围上均有显著差别。
在环形RNA数据的挖掘算法上,赵方庆团队有多篇研究成果发表在Nature Communications,Genome Biology和Briefings in Bioinformatics等国际知名学术期刊上,其中环形RNA的识别算法(CIRI)两年多来的引用次数已超过120次。论文第一作者高远在攻读博士期间获得中科院院长优秀奖(2017)和中国科学院优秀博士论文(2017),现在美国University of Pennsylvania的Perelman School of Medicine进行博士后研究工作。上述研究获得了国家自然科学基金委重大研究计划项目、优秀青年基金项目和中国科学院的经费资助。
论文链接
环形RNA数据挖掘的基本策略

